产品货号:
YTB7109
中文名称:
T3 DNA连接酶
英文名称:
T3 DNA Ligase
产品规格:
150kU|600kU|3000kU
发货周期:
1~3天
产品价格:
询价
本制品是表达纯化获得的一种来源于T3噬菌体、ATP依赖型的双链DNA连接酶。T3 DNA Ligase能够有效催化粘性末端和平末端双链DNA分子的连接以及双链DNA或DNA与RNA杂合双链的缺刻修复。缺刻修复时,完整链为DNA,缺刻链5’端和3’端’分别为DNA-DNA、DNA-RNA、RNA-DNA或RNA-RNA都是可以被T3 DNA Ligase修复的,因此T3 DNA Ligase也可以用于生成DNA-RNA和RNA-DNA融合链接的核酸,以及用于基于DNA模板的RNA连接以形成较长的RNA片段。
T3 DNA Ligase能够催化双链DNA中临近的5'磷酸和3'羟基之间形成磷酸二酯键,且对A/T突出末端的连接效率高于C/G末端。与T4 DNA Ligase相同,在T3 DNA Ligase反应体系中加入PEG6000可以显著提高其对平末端双链DNA的连接效率;而在缺少PEG6000的连接体系中,T3 DNA Ligase对平末端双链DNA分子的连接效率较低。另外,T3 DNA Ligase对NaCl的耐受性是T4 DNA Ligase的2倍,其在1.0M NaCl或KCl的条件下仍可保持95%的活性。因此,在涉及到高离子浓度连接的实验中,T3 DNA Ligase是理想的选择。

限制性内切酶切DNA片段的克隆,PCR产物的克隆连接,双链DNA和接头的连接,线性双链DNA的环化,双链DNA的缺刻修复,定点突变,高盐体系的连接,双链DNA缺刻修复,DNA引导的DNA与RNA连接,DNA引导的RNA连接。

纯化自携带编码T3噬菌体DNA ligase的E.coli重组菌株。

一个单位是指20μL的总反应体积中,25℃条件下,1分钟内使100ng λDNA的HindIII切割片段连接50%所需的酶量。

| 组分 | 150kU | 600kU | 3000kU |
| T3 DNA Ligase(3kU/μL) | 50μL | 200μL | 1mL |
| 2×Reaction Buffer | 500μL | 2mL | 10mL |
保存:-20℃,有效期2年。

10mM Tris-HCl,50mM KCl,1mM DTT,0.1mM EDTA,50% Glycerol,pH7.4@25℃。

2×Reaction Buffer:
132mM Tris-HCl,20mM MgCl2,2mM DTT,2mM ATP,15% PEG6000,pH7.6@25℃。

不含除T3 DNA Ligase之外的其它种类的DNA连接酶,不含内切酶和外切酶,不含RNA酶,不含磷酸酯酶。

在不含有PEG6000的反应体系中65℃孵育10分钟。

- ATP是T3 DNA Ligase发挥催化活性所必需的辅助因子,而不是像E.coli DNA Ligase以NAD作为辅助因子的。因此连接反应体系中一定要保持终浓度为1mM的ATP。本制品提供的2×Reaction Buffer中已经包含了ATP和连接粘末端所需的PEG6000。
- T3 DNA Ligase的催化底物是双链DNA或完整链为DNA的缺刻双链,不能直接用于单链DNA或RNA的连接反应,但可以用于DNA模板引导的单链DNA之间、单链RNA之间或单链DNA与单链RNA之间的连接。
- T3 DNA Ligase的反应体系中含有7.5% PEG6000。如果后续实验体系不兼容PEG6000,可考虑自行配制不含PEG6000的连接反应缓冲液,或者使用T4 DNA连接酶的连接缓冲体系,同时注意添加终浓度为1mM的ATP,但T3 DNA Ligase连接酶在T4 DNA连接酶的连接缓冲体系中的活性会降低约10倍。
- 在不含PEG的反应体系中,T3 DNA Ligase可进行热失活。如果反应体系中含有PEG6000,T3 DNA Ligase不能进行热失活,否则会显著降低后续的转化效率。
- 如果连接体系中需要维持高浓度NaCl,建议使用不含PEG6000的缓冲液。
- 在标准的载体与片段连接的20μL反应体系中,通常需要在25℃下反应30分钟。
- 反应体系所需的超纯水推荐使用无菌无酶超纯水。

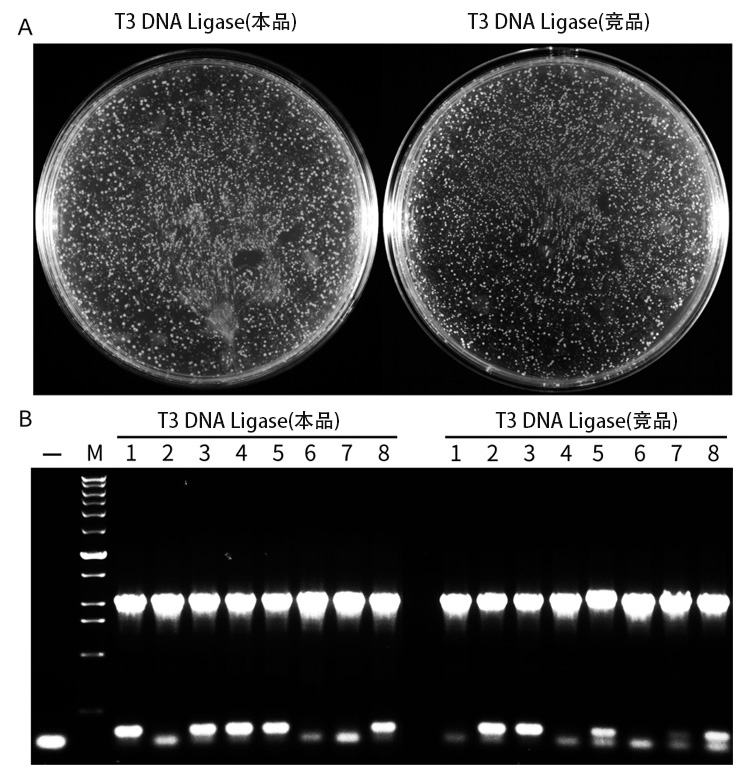
图1.T3 DNA连接酶与N公司的同类产品用于进行双链DNA粘性和平末端连接的效果图。图A为百奥莱博的或N公司的T3 DNA Ligase重组连接产物转化DH5α涂LB平板的实测效果图。利用BalbFusion DNA聚合酶,使用一端带有HindIII酶切识别位点,另一端带有Sma I酶切识别位点的引物对靶基因进行PCR扩增,随后使用HindIII限制性内切酶/SmaI限制性内切酶对1kb的PCR产物进行双酶切,获得一端带有粘性末端,另一端带有平末端的双链线性DNA分子,作为T3 DNA Ligase催化连接反应的底物。在20μL反应体系(66mM Tris-HCl,10mM MgCl2,1mM DTT,1mM ATP,7.5% PEG6000,pH7.6@25℃)中,加入50ng经PCR扩增及Hind III/Sma I双酶切产生的两端分别带有粘性末端和平末端的双链DNA片段,和50ng经Hind III/Sma I双酶切线性化的pUC18载体(待插入DNA片段与pUC18载体的摩尔比为3:1),再加入10μL的2×Reaction Buffer以及1μL的本制品或N公司的T3 DNA Ligase,然后用水补至20μL,25℃孵育30分钟进行连接。反应结束后,取5μL连接产物转化DH5α感受态细胞。图B为菌落PCR鉴定T3 DNA Ligase重组连接构建得到的克隆。实验结果表明,本制品与N公司的产品具有相当的连接粘性末端和平末端双链DNA的效果。菌落PCR鉴定使用的是pUC18载体的通用测序引物:M13 forward sequencing primer (5'-GTAAAACGACGGCCAGT-3')和M13 reverse sequencing primer (5'-CAGGAAACAGCTATGAC-3')。本图仅供参考,实际检测效果可能有所不同。

- 参考下表在冰浴中配制反应体系(以20μL体系为例)。
成分 用量 2×Reaction Buffer 10μL 载体DNA XμL(0.020pmol) 待插入DNA片段 YμL(0.060pmol) 超纯水 (9-X-Y)μL T3 DNA Ligase(3kU/μL) 1μL 总体积 20μL - 建议按照插入DNA片段与线性化载体摩尔比3:1的比例进行连接反应。
- T3 DNA Ligase建议最后添加。
- 建议按照插入DNA片段与线性化载体摩尔比3:1的比例进行连接反应。
- 移液器吹打混匀,低速离心使粘附在管壁上的液体沉降至管底。
- 反应条件:25℃(或室温)孵育15~30分钟。
- 反应完成后需将连接产物立即置于冰上,取5μL左右连接产物转化到50μL感受态细胞中。剩余样品可保存于-20℃。
- 不能进行热失活,热失活会显著降低连接产物的转化效率。
- 为检测连接效率,也可将反应后的产物进行琼脂糖凝胶或聚丙烯凝胶电泳,拍照观察并分析连接效果。如果需要从琼脂糖凝胶中回收DNA样品,推荐使用琼脂糖凝胶DNA回收试剂盒。
- 不能进行热失活,热失活会显著降低连接产物的转化效率。

- T3 DNA Ligase可以和不含PEG6000的缓冲液一起使用吗?
可以。如果实验体系中不能加入PEG6000,推荐自行配制仅去除了PEG6000的2×Reaction Buffer。 - 使用T3 DNA Ligase进行连接反应时,有哪些潜在的因素会导致转化失败?
有以下因素会导致连接反应失败:- 反应体系中缺少ATP或Mg2+会导致连接失败。缓冲液中的ATP,保存时间过长可能会逐渐降解导致此问题的发生。建议使用新鲜提供的缓冲液或适量补充ATP到反应体系中,以保证连接效率。
- 反应体系中存在高盐或EDTA会导致连接失败。建议纯化连接底物,去除干扰。
- CIP、BAP或SAP等磷酸酶在去磷酸化过程中没有完全失活。建议按照推荐的步骤完全去除磷酸酶。
- 反应体系中DNA的浓度过高会导致只产生线性DNA。建议连接体系中DNA的总浓度保持在1~10μg/mL的范围内。
- 加入太多的连接产物转化到感受态细胞中会导致转化的失败。建议加入1~5μL的连接产物转化到50μL的感受态细胞中。
- 含有PEG6000的情况下过长时间的连接,会逐渐产生抑制转化的大片段DNA,会降低转化效率。
- 连接产物在电穿孔前没有进行纯化。缓冲液中存在的盐和PEG6000均会抑制电穿孔实验。建议使用纯化柱对连接产物进行纯化,以尽可能去除缓冲液。
- 空载体酶切不完全,会导致获得的克隆基本都是空载体而缺少含有插入片段的目的克隆。
- 反应体系中缺少ATP或Mg2+会导致连接失败。缓冲液中的ATP,保存时间过长可能会逐渐降解导致此问题的发生。建议使用新鲜提供的缓冲液或适量补充ATP到反应体系中,以保证连接效率。
- 在解决转化效率问题时还应考虑哪些因素?
- 感受态细胞不能存活或转化效率过低。建议使用新的感受态细胞。
- 连接的DNA是否含有大肠杆菌拮抗的反向重复序列或串联重复序列。
- 插入的DNA片段如果来自哺乳动物或者植物,可能含有能够被多种大肠杆菌株所降解的甲基化胞嘧啶。建议使用mcrA、mcrBC和mrr缺乏的大肠杆菌株。
- 构建的载体过大(>10kb),不能使用化学转化的方式,建议使用电穿孔转化方式。
- 感受态细胞不能存活或转化效率过低。建议使用新的感受态细胞。
- 在限制性内切酶消化过程中存在哪些问题会导致T3 DNA Ligase的连接反应或后续的转化失败?
- 酶切效率不高,没有实现完全酶切。如果切割发生在一个PCR片段的末端,需要确保有足够的酶切保护碱基,建议在酶切位点外侧额外增加6个碱基。并建议使用对照底物测试限制酶的活性。
- 限制性内切酶没有完全失活。如果限制性内切酶不能热失活,可以纯化DNA以尽可能地去除限制性内切酶。
- 限制性内切酶切割DNA片段或载体时产生了星活性。建议凝胶电泳检测DNA,适当减少限制性内切酶的使用量或减少酶切反应时间。
- DNA或限制性内切酶中含有破坏DNA片段末端的核酸外切酶或磷酸酶时,建议纯化DNA。
- 酶切效率不高,没有实现完全酶切。如果切割发生在一个PCR片段的末端,需要确保有足够的酶切保护碱基,建议在酶切位点外侧额外增加6个碱基。并建议使用对照底物测试限制酶的活性。
- 使用T3 DNA Ligase时应加入多少DNA?
为促进环化DNA连接产物的形成,提高转化效率,加入的总DNA浓度应在1~10μg/mL之间,以实现有效连接。同时建议按照插入DNA片段与线性化载体摩尔比3:1的比例加入到反应体系中。摩尔比低于2:1会降低连接效率;摩尔比高于6:1会导致多个片段的插入。如果底物DNA的浓度无法确定,可尝试多种比例的连接。 - T3 DNA连接酶对盐离子是否比T4 DNA连接酶具有更高的耐受性?
是的。连接反应中T3 DNA Ligase能够耐受250~300mM的盐浓度。同时请注意,在PEG存在的连接体系中,DNA在高盐条件下会沉淀,并抑制连接反应的进行。 - T3 DNA Ligase能否被热失活?
可以。在不含有PEG的反应体系中65℃孵育10分钟能够将T3 DNA Ligase热失活;但在含有PEG的反应体系中,T3 DNA Ligase可以进行热失活,但会影响后续的转化效率。因此在后续用于转化的情况下,不建议进行热失活。 - 当使用T3 DNA连接酶时,应该使用什么对照来测试细胞和DNA?
- 对每个对照均使用相同的DNA浓度,通常每次转化0.1~1.0ng的DNA。
- 在感受态细胞中转化未经酶切线性化的载体并均匀涂布到不含有/含有抗生素的培养基平板上,可检测细胞活力和质粒的抗生素耐受性。
- 检测线性化后的载体是否含有未被完全切割的质粒背景。将线性化后的载体转化感受态细胞,获得的克隆数目应小于步骤a中克隆数目的10%。
- 使用切割并重新连接的质粒检测连接酶的活性和DNA末端的完整性。将切割并修饰后的载体转化感受态细胞,获得的克隆数目应接近步骤a中克隆数目。
- 使用切割,磷酸化并重新连接的质粒检测由于磷酸化酶处理不完全所导致的背景。将切割,磷酸化并重新连接的质粒转化感受态细胞,获得的克隆数目应小于步骤a中克隆数目的10%。
相关搜索:T3 DNA连接酶,DNA连接酶,Ligase,T3 DNA Ligase



